Resultados
En esta página se detallarán los resultados de las distintas etapas del proyecto:
Binarización de las distintas especies de colonias
En la Figura 1 se puede observar el resultado del procesamiento de las imágenes de las placas de Petri para un conjunto de 5 imágenes de cada especie de colonias. En la misma se puede observar cómo el algoritmo se desempeña de manera correcta para la mayoría de las imágenes, en el caso de C.albicans, se trata de colonias muy pequeñas por lo que la detección y el procesamiento adquieren otro grado de complejidad. El resto de las colonias aparecen bien definidas y no existe presencia de texto.

Conteo para las distintas especies de colonias
Para poder calcular métricas relacionadas a el conteo de las especies de colonias se definieron ciertas métricas. Para contabilizar una colonia como correcta primero calculé el área de intersección o el IOU entre el cuadrado delimitador original y el que el algoritmo definió, si existe solapamiento es debido a la correcta identificación de la colonia. A partir de esto se pueden obtener las siguientes métricas:
- Verdaderos positivos: Aquellas colonias que fueron identificadas correctamente como colonias.
- Falsos positivos: Aquellas colonias que fueron identificadas como colonias pero que no lo eran.
- Falsos negativos: La cantidad de colonias que tenían un cuadrado delimitante pero no lo detecté.
A partir de estos valores puedo calcular la precisión, el recall y el F1 Score. La precisión mide de las colonias que el algoritmo predijo como colonias, cuántas son verdaderamente colonias. Por otro lado, el recall mide de todos las colonas que realmente eran colonias, cuántas predijo correctamente el algoritmo. Por último, el F1 score, es una medida del desempeño del modelo y se utiliza tanto la precisión como el recall para su cálculo. Este es el promedio armónico de las anteriores, lo que da como resultado un balance entre las mismas.
En la siguiente tabla se obtienen los promedios para cada una de las especies de colonias en la etapa de detección:
| Class | Precision | Recall | F1 Score |
|---|---|---|---|
| E.coli | 0.988889 | 0.955556 | 0.971466 |
| P.aeruginosa | 0.938333 | 0.846667 | 0.882888 |
| B.subtilis | 0.670707 | 0.779770 | 0.717523 |
| S.aureus | 0.750000 | 0.630952 | 0.680769 |
La especie C.albicans no pudo ser detectada debido a la similitud de las colonias con el ruido detectado. De la tabla anterior se puede observar que en la clase que mejor se desempeña la detección es E.coli, esto puede deberse a la uniformidad de las colonias de E.coli, a diferencia de las otras especies dónde las colonias son menos uniformes y con características mas variables.
Una fuente de error en los falsos positivos es la presencia de ruido o artefactos en las imágenes binarizadas, estos son producto del procesamiento inicial que luego no son descartadas correctamente en la etapa de conteo.
Detección de especies a partir de colonias
Para esta parte del proyecto se utilizaron diferentes combinaciones de características y se probaron con los modelos anteriormente mencionados. Debido a la poca diferencia entre las colonias y la gran variabilidad dentro de cada especie, encontrar las características que separen correctamente las colonias no es una tarea fácil. Se probaron diversas variaciones en las características utilizadas, como así también teniendo en cuenta las características de todo el cuadrado delimitador de la colonia o sólo del área que pertenecía a la colonia, para saber esto se utilizaron los procedimientos de la parte anterior pero aplicados a solo el cuadrado delimitador y no toda la imagen de la placa de Petri.
En la siguiente tabla se muestran los resultados para SVM y Random Forest teniendo en cuenta todas las características incluyendo el cuadro delimitador y el área de la colonia dentro del mismo. En estas iteraciones se utilizó una cantidad de 2564 colonias.
| Model | Profunidad/Kernel | Accuracy |
|---|---|---|
| RF | Profundidad: 5 | 0.46 |
| RF | Profundidad: 10 | 0.50 |
| RF | Profundidad: 15 | 0.49 |
| RF | Profundidad: 20 | 0.49 |
| RF | Profundidad: 25 | 0.48 |
| SVM | linear | 0.46 |
| SVM | poly | 0.33 |
| SVM | rbf | 0.31 |
| SVM | sigmoid | 0.30 |
Se puede ver cómo el mejor clasificador fue RandomForest con 0.49 de accuracy, si de este modelo se analiza el valor de recall, precisión y F1 para cada categoría se obtiene la siguiente tabla:
| Class | Precision | Recall | F1-Score |
|---|---|---|---|
| E.coli | 0.37 | 0.11 | 0.17 |
| S.aureus | 0.50 | 0.56 | 0.53 |
| B.subtilis | 0.41 | 0.53 | 0.47 |
| P.aeruginosa | 0.29 | 0.38 | 0.33 |
| C.albicans | 0.64 | 0.41 | 0.50 |
| Accuracy | 0.43 |
Vemos que la precisión es mas alta en S.aureus y C.alibicans, esto va de la mano a que estas colonias tienen los colores más oscuros y distinguibles entre sí en comparación a las otras tres especies que el color de las colonias es muy parecido.
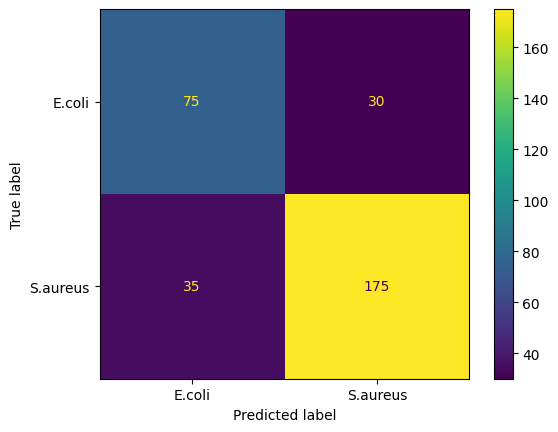
Si en cambio tomamos únicamente dos colonias para utilizar en la clasificación, el desempeño del mejor modelo cambia como se puede ver a continuación:
| Class | Precision | Recall | F1-Score |
|---|---|---|---|
| E.coli | 0.69 | 0.70 | 0.69 |
| S.aureus | 0.85 | 0.84 | 0.84 |
| Accuracy | 0.79 |
En este caso aumenta el desempeño en general como se observa en la accuracy pero también la precisión dentro de cada categoría.
En la Figura 2 se puede observar la matriz de confusión para el modelo mencionado.
Verificación de todo el pipeline
En última instancia se implementó un algoritmo para a partir de una imagen de una placa de petri, se procediera a binarizar la imagen, detectar las colonias y clasificarlas. En este caso el accuracy de la clasificación fue de 0.56%, un valor bajo que puede ser atribuido a el mal desempeño de los clasificadores entrenados.